分子表示ソフト Detrial
Detrial は,タンパク質を中心とする分子構造の解析ツールを企図して試験的に開発しているフリー(オープンソース)ソフトウェアです.
タンパク質の構造解析ソフトを中心とした検知アルゴリズムの勉強と実装を志向しています.
- 分子,タンパク質(アミノ酸配列)の検知
- 2 個の描画領域を有しており,それぞれから選択したペプチドの配列アラインメント,構造アラインメント
- アミノ酸/非アミノ酸,選択分子/非選択分子,選択アミノ酸/その他を区別した描き分け
- 中心からの距離による色分け,特定のアミノ酸の表示
- 近傍の原子,接触している原子の検知
- 水素結合や電荷の検知
- k-平均法によるクラスター検知(ドメイン検知)
- 原子の座標の固有ベクトル計算(分子の概形)
現在は,GUI 部分も含め試験的な機能やデータ構造を追加中であり,不具合を多く含む可能性があります.
2024 年には,バージョン 2 の開発を再設計段階から開始する予定です.
実行イメージ
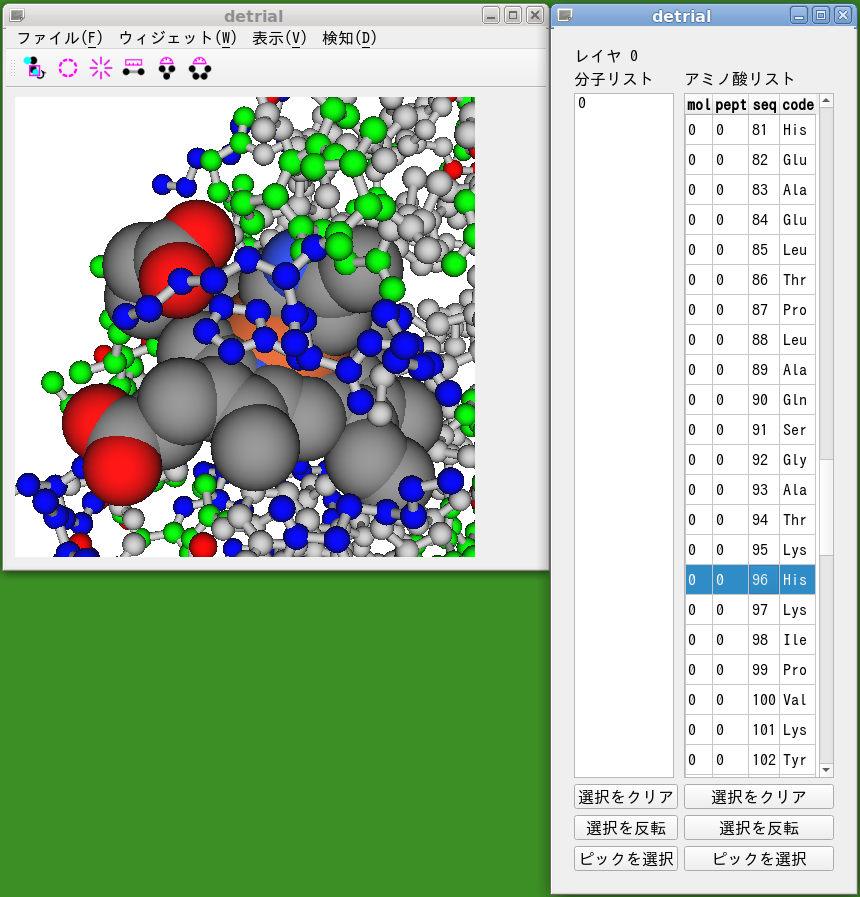
画像は,ヘモグロビンのヘム近傍拡大イメージです.
ヘム鉄近傍のヒスチジンを選択した結果,画面右側にあるアミノ酸リストのヒスチジンが 1 個反転表示されています.
作業の概略を下に記します.
資料としたファイルは,Builcule を使って 1mbn.pdb から単離して作成しました.
アミノ酸/非アミノ酸に基づく描き分け機能を利用しています.
アミノ酸は「棒球リボン」様式(棒球モデルとリボンモデルを重ね書きしたもの)で表示し,棒球モデルはアミノ酸の性質に基づいて色分けし,リボンモデルは二次構造に基づいて着色しています.
非アミノ酸を空間充填様式で表示し CPK 色に着色しました.
ズーム機能でヘム近傍を拡大しました.
鉄原子の近くにあるヒスチジンをピックし,アミノ酸セレクタで選択しました.
使用法
メニュー
[ファイル(F)]
[開く(O)]:XYZ 形式,PDB 形式,PDBx/mmCif 形式,および独自形式である BCL 形式に対応しています[Open Babel でインポート]:リストビューで表示される対応形式を選択したら,ファイル選択ダイアログボックスが表示されます
[保存(S)]:XYZ 形式,PDB 形式,および独自形式である BCL 形式に対応しています.なお,PDB 形式については一部の機能が未完成です
[Open Babel でエクスポート]:リストビューで表示される対応形式を選択したら,ファイル選択ダイアログボックスが表示されます
[FASTA 形式で出力(F)]:検知したすべてのペプチドをマルチ FASTA 形式で出力します
[画像として保存(I)]:ウィンドウを PNG 形式の画像として保存します
[終了(Q)]
[ウィジェット(W)]
[レイヤ 0]:1 個めの分子(系)を開く領域を使います[レイヤ 1]:2 個めの分子(系)を開く領域を使います
[レイヤ 01]:レイヤ 0 とレイヤ 1 を同時に使います
[セレクタを表示]:分子やアミノ酸を選択するためのウィジェットを表示します
[編集(E)]
[ピックした原子を重ね合わせ(平面上)]:平面上に位置する 1〜3 原子を重ね合わせます[ピックした原子を重ね合わせ(非平面上)]:最小二乗法でピックした原子を重ね合わせます
[センタリング]:系の中心と半径を再設定し,全分子を表示します
[表示(V)]
[空間充填]:原子を球で表します.球の半径はファンデルワールス半径です--[CPK]:原子を CPK 様式で着色します
--[分子]:原子を分子ごとに着色します
--[アミノ酸]:原子をアミノ酸の性質にしたがって着色します
[棒球 + CPK色]:原子を球で,共有結合を棒で表します.原子については CPK 様式で着色します
[リボン + 二次構造色]:ペプチド主鎖をリボンで表し,α-ヘリックスを赤色に,β-シートを黄色に着色します
[描き分け]
--[アミノ酸/非アミノ酸]
--[選択分子/非選択分子]
--[選択アミノ酸/その他]
[電荷の簡易計算]:原子を空間充填モデルで表示し,正電荷を有する原子は青色に,負電荷を有する原子は赤色に着色します
[電荷のインポート]:別途作成した原子の電荷を記したファイルをインポートして表示する機能です
[検知(D)]
[行順位を指定してピック]:開いているファイルの原子の順番(1 から始まります)を指定してピックします[ピックした原子の近傍アミノ酸を選択]:ピックした原子からの距離を指定して,アミノ酸セレクタのアミノ酸を選択します(近傍の検知)
[選択した分子の近傍アミノ酸を選択]:分子セレクタで選択した分子からの距離を指定して,アミノ酸セレクタのアミノ酸を選択します(近傍の検知)
[選択したアミノ酸の近傍アミノ酸を選択]:アミノ酸セレクタで選択した分子からの距離を指定して,アミノ酸セレクタのアミノ酸を選択します(近傍の検知)
[選択した 2 分子間の近傍原子を検知]:分子セレクタで選択した 2 分子について,指定した距離以内の原子を空間充填で,他は針金で表示します(近傍の検知)
--[分子別に色分け]:空間充填で表す原子の色を,分子別に赤色と青色に塗り分けます
--[電荷を計算して着色]:空間充填で表す原子の色を,電荷を計算して着色します
[アラインメント]:アラインメントを作成し,タンパク質の重ね合わせその他の操作をおこないます
[ドメイン]:k-平均法でクラスター(タンパク質ではドメイン)を検知します.想定するクラスター数を指定して検知を実行します(実験的な機能:ドメイン)
[接触原子]:ファンデルワールス距離近傍の原子を検知します(実験的な機能:接触原子)
[座標分布]:原子のおおまかな分布方向と分布幅を計算します(実験的な機能:座標分布)
[深度]:中心からの距離を色のグラデーションで表現します(実験的な機能:深度)
[水素結合]:距離と角度を指定して水素結合を検知します(実験的な機能:水素結合)
[アミノ酸の種類を指定]:指定(複数可)されたアミノ酸を球で,他の部分は棒で表示します(実験的な機能:アミノ酸の種類を指定)
[コンタクトマップ]:アミノ酸の α-炭素間の距離に基づくコンタクトマップです(実験的な機能:コンタクトマップ)
レイヤ
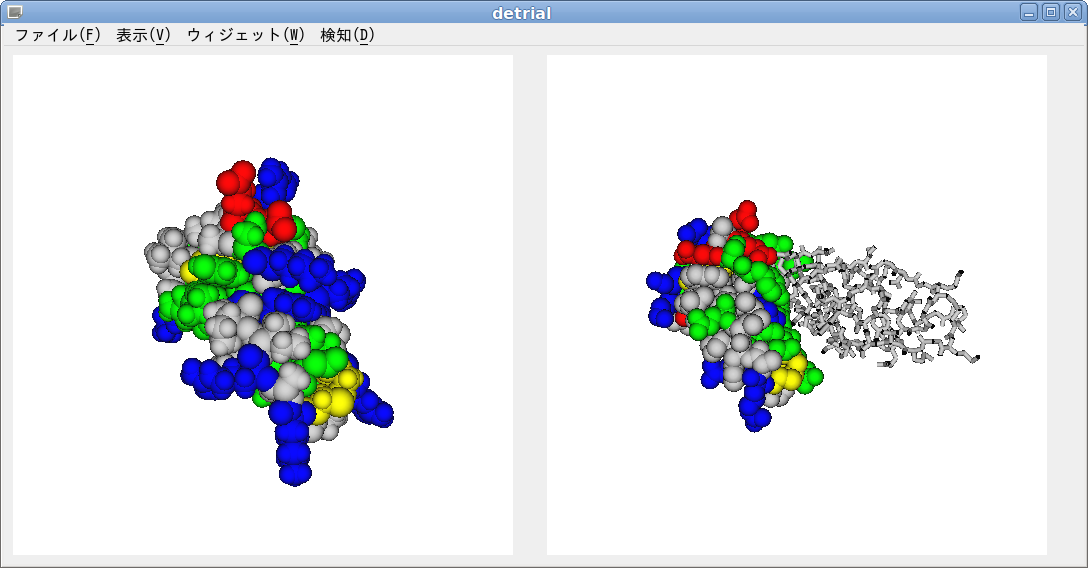
Detrial は,独立に 2 個の系(== 2 個のファイル)を処理でき,レイヤ 0,およびレイヤ 1 と名付けています.
これらは,[ウィジェット(W)]-[レイヤ 0] / [レイヤ 1] で利用できます.
両方のレイヤを同時に表示することも可能で,その場合は [ウィジェット(W)]-[レイヤ 01] とします.
画像は,2 個のレイヤを同時に表示している例で,アラインメントの結果に従ってタンパク質を重ね合わせたところです.
マウスとキーボードによる分子の回転と平行移動
処理上優先している順に記します.
(1) 両方のレイヤで分子を開いておき,A キーを押しながらマウスをドラッグすると両方のレイヤの分子が同じ角度だけ回転します.
(2) X キーを押したまま同じ操作をすると,マウスをドラッグすると視点が平行移動します(全分子が平行移動するように見えます)
(3) Z キーを押しながらマウスホイールを回転するとズームイン/ズームアウトします.
(4) セレクタで分子を選択し,水平ツールバーの [選択分子ごと] ボタン![]() を押した状態にして
を押した状態にして
・右ドラッグすると選択した分子が平行移動します.
・中(== 左右)ドラッグすると選択肢た分子のうち,番号が最小のものがその場で回転します.
(5) 左ドラッグすると分子が回転します.
水平ツールバー
 選択分子のみ移動・回転:このボタンを押し込んだ状態にすると,セレクタで選択した分子の移動や回転がおこなえます
選択分子のみ移動・回転:このボタンを押し込んだ状態にすると,セレクタで選択した分子の移動や回転がおこなえます ピックモード:このボタンを押し込んだ状態にすると,ピック操作を受け付けます
ピックモード:このボタンを押し込んだ状態にすると,ピック操作を受け付けます ピックを解除:ピックが解除されて,赤紫に表示されている原子の色は元に戻ります
ピックを解除:ピックが解除されて,赤紫に表示されている原子の色は元に戻ります 距離を測定:ピックした 2 原子間の距離をポップアップ表示します
距離を測定:ピックした 2 原子間の距離をポップアップ表示します 角度を測定:ピックした 3 原子が成す角度をポップアップ表示します
角度を測定:ピックした 3 原子が成す角度をポップアップ表示します 二面角を測定:ピックした 4 原子が成す二面角をポップアップ表示します
二面角を測定:ピックした 4 原子が成す二面角をポップアップ表示します
ピックによる原子の選択
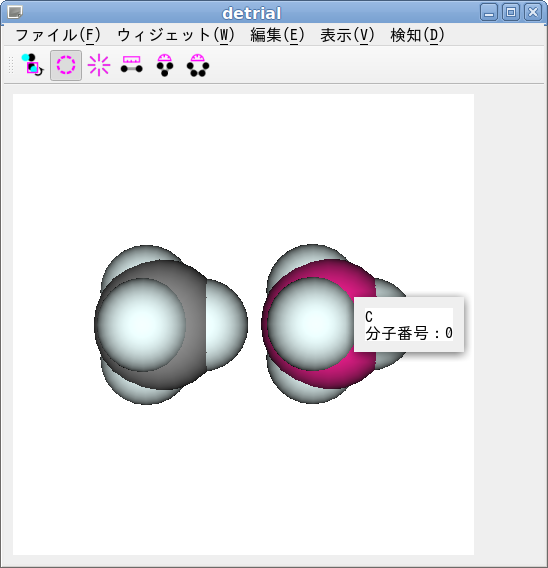
画像は,2 個並んだメタンのうち,1 個の炭素をピックしたところです.
水平ツールバーに配置している![]() ピックモードボタンは,トグルボタンになっています.
ピックモードボタンは,トグルボタンになっています.
これを押し込んだ状態で,球で表示されている原子を左クリックすると,その原子の大きさが空間充填様式になり,色が赤紫色に変わります.
この操作をピックと称します.
ピックの際,その原子の情報がポップアップウィンドウに表示されます.
ツールバー左端のボタンを押し込まれていない状態にすると,ピックできなくなります.デフォルトではこの状態です.
セレクタによる分子とアミノ酸の選択
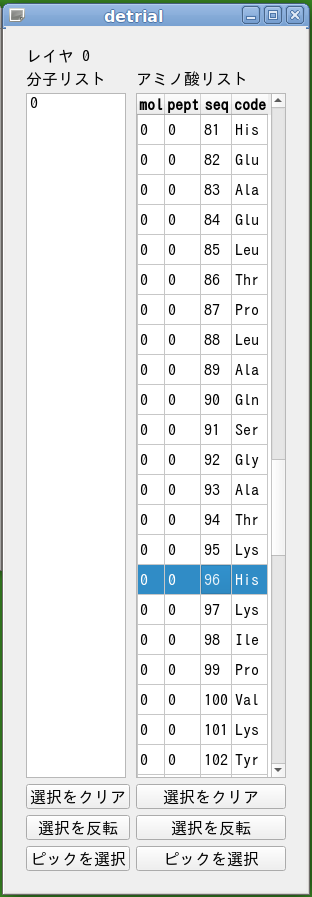
メニューで [ウィジェット(W)]-[セレクタを表示] とすれば,分子の番号や,ペプチドの番号,さらにアミノ酸の3文字コードが並んだセレクタが表示されます.
実行イメージに,セレクタが表示されています.
マウスでセレクタ上の分子またはアミノ酸をクリックすれば,その行が反転表示されます.これは選択状態を表します.
Ctrl キーや Shift キーを押しつつクリックすることで,マウスの動作を変更できます.
メニューの [表示(V)]-[選択モード] と組み合わせて使うと,選択した分子やアミノ酸の系全体での位置がわかります.
ウィジェット底部に選択の状態を変更するためのボタンを置いています.
アラインメントを作成すると,アラインメントされたアミノ酸が自動的に選択されます.
ビルドとインストール
- ビルドには,C++ コンパイラ + Qt + Eigen + Open Babel ライブラリが必要です(下記「開発環境」参照)
- まず,libbuilcule-1 をインストールし,その後で Detrial をビルドしてください
ソースファイル
libbuilcule-1_20220829.tar.bz2:共有ライブラリです.スケジュールの都合がつかなかったので,旧版の libbuilcule-1 を利用しています
detrial-1_b15.tar.bz2:本体.あらかじめ上の libbuilcule をインストールしておく必要があります
開発環境
ビルドには,C++ コンパイラ + Qt + Eigen + Open Babel ライブラリが必要です.
下の表は,私がインストールしているパッケージです.
プログラミングに,開発環境の練習記録があります.
| パッケージ名(バージョン) | 注 |
| g++ (10.2.1) | GNU C++ コンパイラ |
| qtbase5-dev (5.15.2) | GUI ライブラリ.自動的に Makefile 作成ツール qt5-qmake もインストールされます |
| libeigen3-dev (3.3.9) | 線形代数用のライブラリ.分子の三次元座標の処理 |
| libopenbabel-dev (3.1.1) | ライブラリ版 Open Babel |
libbuilcule のビルド
libbuilcule-1_20220829.tar.bz2 を解凍して生成するディレクトリに移動します.
~$ tar -xjvf libbuilcule-1_20220829.tar.bz2
~$ cd libbuilcule-1_20220829
Makefile を作成します.
~/libbuilcule-1_20220829$ qmake
続いてビルド.
~/libbuilcule-1_20220829$ make
ビルドが成功したら実行ファイル libbuilcule.so.1.0.0 およびそれへのシンボリックリンクがいくつか生成します.
libbuilcule のインストール
ビルドに成功したら,
~/libbuilcule-1_20220829$ sudo make install
/usr/local/include/builcule/ にヘッダファイルが,/usr/local/lib/builcule/ にライブラリがインストールされます.
共有ライブラリのキャッシュを更新してください.
~/libbuilcule-1_20220829$ sudo ldconfig
libbuilcule のアンインストール
アンインストールは,qmake を実行した後の状態で,
~/libbuilcule-1_20220829$ sudo make uninstall
とします.
バージョンアップでファイルの増減や名前の変更をする場合があるので,ディレクトリと一部のファイルが残ったままになるかもしれません.
そのような場合は,
~/libbuilcule-1_20220829$ sudo rm -rf /usr/local/lib/builcule
~/libbuilcule-1_20220829$ sudo rm -rf /usr/include/lib/builcule
として削除してください.
Detrial のビルド
detrial-1_b15.tar.bz2 を解凍して生成するディレクトリに移動します.
~$ tar -xjvf detrial-1_b15.tar.bz2
~$ cd detrial-1_b15
Makefile を作成します.
~/detrial-1_b15$ qmake
続いてビルド.
~/detrial-1_b15$ make
ビルドが成功したら実行ファイル detrial が生成します.
Detrial のインストール
ビルドに成功したら,
~/detrial-1_b15$ sudo make install
で /usr/local/bin/ にインストールされます.
実行
インストールしてあれば,任意のディレクトリから,
detrial &
で Detrial が起動します.
アンインストール
アンインストールは,qmake を実行した後の状態で,
~/detrial-1_b15$ sudo make uninstall
とします.
現状と予定
不具合
- 表示様式を変更しても反映されない場合は,マウスで分子を回転させるなどの操作をおこなってみてください
- 構造をクリーンアップする機能を実装していないので,タンパク質のデータベースからダウンロードしたファイルを開くと異常終了する場合があります(必要に応じて Builcule を使ってクリーンアップして下さい)
Detrial リリース履歴
2022 年 8 月 29 日 :共有ライブラリのアップデートです.ファイルに記述された元素記号がすべて大文字であっても読み込めるようにしました.
2022 年 8 月 10 日 : β-15 リリース.電荷を簡易計算して表示する機能を復活させました.
2022 年 8 月 9 日 : β-14 リリース.電荷を簡易計算して表示する機能に代えて,電荷を記したファイルをインポートして表示する機能を作成しました.
2022 年 7 月 1 日 : β-13 リリース.コンタクトマップの表示機能を追加しました.
2022 年 6 月 16 日 : β-12 リリース.ウィンドウのクローズイベントの処理が誤っていたので修正しました.
2022 年 6 月 15 日 : β-11 リリース.アミノ酸の種類を指定して検知する機能を追加しました.
2022 年 6 月 14 日 : β-10 リリース.水素結合を検知する機能を追加しました.
2022 年 6 月 13 日 : β-9 リリース.中心からの距離を色のグラデーションで表現する機能を追加しました.
2022 年 6 月 12 日 : β-8 リリース.原子分布の固有ベクトルを表示する機能を追加しました.
2022 年 6 月 9 日 : β-7 リリース.近傍原子の検知機能を追加しました.ドメイン検知機能を修正しました.いくつかのバグを修正しました.
2022 年 4 月 18 日 : β-6 リリース.タンパク質側鎖の検知のバグ,タンパク質のアラインメントのバグを修正しました.
2022 年 2 月 27 日 : β-5 リリース.アンドゥ,リドゥ機能を追加しました.その他,電荷の扱いやバグの修正等,いくつかの変更を加えています.
2022 年 2 月 10 日 : β-4 リリース.ファイルの入出力に Open Babel の機能を追加しました.原子を指定する重ね合わせ機能を作成しました.
2022 年 1 月 23 日 : β-3 リリース.ペプチドの重ね合わせに関する機能を修正しました.
2021 年 12 月 26 日 : β-2.1 リリース.ペプチドを含むファイルを連続して開くと異常終了するバグを修正しました.
2021 年 12 月 12 日 : β-2 リリース.Builcule-9 との共通部分を共有ライブラリとしました.
2021 年 5 月 26 日 : β-1 リリース
2021 年 3 月 12 日 : α-1 リリース.